ロジスティック回帰は線形分離可能なデータの境界線を学習によって見つけてデータの分類を行なう手法です。
特徴としては境界線が直線になることです。
また、データがクラスに分類される確率も計算することが可能です。
欠点としては教師データが線形分離可能でないと分類ができないということです。
また、教師データから学習した境界線はクラスの端にあるデータのすぐそばを通るようになるため、一般化した境界線になりにくい(汎化能力が低い)ことも欠点です。
実装
ロジスティック回帰モデルはscikit-learnライブラリのlinear_modelサブモジュール内にLogisticRegression()として定義されています。
ロジスティック回帰モデルを使って学習する場合、次のようなコードを書いてモデルを呼び出します。
# パッケージからモデルを呼び出す
from sklearn.linear_model import LogisticRegression
# モデルを構築する
model = LogisticRegression()
# モデルに学習させる
# train_data_detailはデータのカテゴリーを予測するために使う情報をまとめたもの
# train_data_labelはデータの属するクラスのラベル
model.fit(train_data_detail, train_data_label)
# モデルに予測させる
model.predict(data_detail)
# モデルの予測結果の正解率
model.score(data_detail, data_true_label)
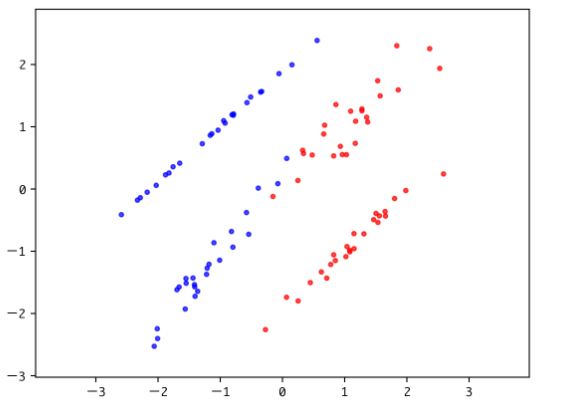
今回は座標によって属するクラスを識別しているため、グラフでモデルがどのような境界線を学習したのか見ることができます。
境界線は直線なので、y = ax+b の形で表現されます、以下のXi, Yはその直線を求めている過程になります。
グラフの視覚化にはmatplotlibライブラリを使います。
# パッケージをインポート
import numpy as np
import matplotlib
import matplotlib.pyplot as plt
# ページ上で直接グラフが見られるようにするおまじない
%matplotlib inline
# 生成したデータをプロット
plt.scatter(X[:, 0], X[:, 1], c=y, marker=’.’,
cmap=matplotlib.cm.get_cmap(name=’bwr’), alpha=0.7)
# 学習して導出した識別境界線をプロット
# model.coef_はデータの各要素の重み(傾き)を、
# model.intercept_はデータの要素全部に対する補正(切片)を表す。
Xi = np.linspace(-10, 10)
Y = -model.coef_[0][0] / model.coef_[0][1] * \
Xi – model.intercept_ / model.coef_[0][1]
plt.plot(Xi, Y)
# グラフのスケールを調整
plt.xlim(min(X[:, 0]) – 0.5, max(X[:, 0]) + 0.5)
plt.ylim(min(X[:, 1]) – 0.5, max(X[:, 1]) + 0.5)
plt.axes().set_aspect(‘equal’, ‘datalim’)
# グラフにタイトルを設定する
plt.title(“classification data using LogisticRegression”)
# x軸、y軸それぞれに名前を設定する
plt.xlabel(“x-axis”)
plt.ylabel(“y-axis”)
plt.show()
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 |
# パッケージをインポート import numpy as np import matplotlib import matplotlib.pyplot as plt from sklearn.linear_model import LogisticRegression from sklearn.model_selection import train_test_split from sklearn.datasets import make_classification # ページ上で直接グラフが見られるようにするおまじない %matplotlib inline # データの生成 X, y = make_classification(n_samples=100, n_features=2, n_redundant=0, random_state=42) train_X, test_X, train_y, test_y = train_test_split(X, y, random_state=42) # 以下にコードを記述してください # モデルの構築 model = LogisticRegression(random_state=42) # train_Xとtrain_yを使ってモデルに学習させる model.fit(train_X, train_y) # test_Xに対するモデルの分類予測結果 pred_y = model.predict(test_X) # 生成したデータをプロット plt.scatter(X[:, 0], X[:, 1], c=y, marker='.', cmap=matplotlib.cm.get_cmap(name='bwr'), alpha=0.7) # 学習して導出した識別境界線をプロット Xi = np.linspace(-10, 10) Y = -model.coef_[0][0] / model.coef_[0][1] * \ Xi - model.intercept_ / model.coef_[0][1] plt.plot(Xi, Y) # グラフのスケールを調整 plt.xlim(min(X[:, 0]) - 0.5, max(X[:, 0]) + 0.5) plt.ylim(min(X[:, 1]) - 0.5, max(X[:, 1]) + 0.5) plt.axes().set_aspect('equal', 'datalim') # グラフにタイトルを設定する plt.title("classification data using LogisticRegression") # x軸、y軸それぞれに名前を設定する plt.xlabel("x-axis") plt.ylabel("y-axis") plt.show() |